如何使用trim_galore对NGS数据质量进行过滤,相信很多没有经验的人对此无能为力。为此,本文总结了问题产生的原因和解决方法,希望大家可以通过这篇文章来解决这个问题。
Cutadapt软件可以过滤NGS数据的质量,FastQC软件可以检查NGS数据的质量分布,trim_galore将这两个软件打包在一起,使用起来更加方便。
软件将在以下四个步骤中处理数据
1. 去除reads 3’端的低质量碱基
illumina平台测序数据,通常3’端质量较差。Trim_galore将首先在3’端过滤掉低质量的碱基,本质上是调用cutadapt的质量过滤算法。下图显示了过滤前后的基础质量分布。
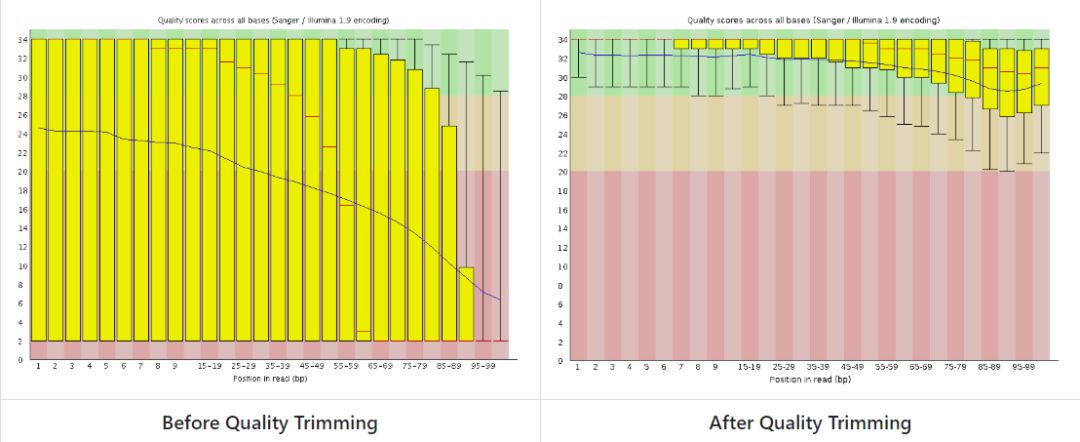
可以看出,过滤掉低质量的碱基后,序列的整体质量显著提高。
00-1010在过滤掉低质量的碱基后,trim_galore将调用cutadapt在读取的3’端找到适配器序列并将其移除。通常,我们需要指定相应的适配器序列。如果没有规范,trim_galore会自动找到以下三种类型的适配器。
illumina a : agatcggaagagc
SmallRNA:TGGAATTCTCGG
Nextera:CTGTCTCTTATA默认读取前一百万个序列,通过这一百万个序列判断适配器属于以上三种类型中的哪一种,然后删除。如果不希望软件自动判断,也可以通过-illumina、-nextera、-small _ RNA参数指定对应的适配器类型。
00-1010经过以上两个步骤,有可能剩余的序列长度很短,而这个短序列也将被删除。默认情况下,如果序列长度小于20bp,序列将被丢弃。
2. 去除adapter序列
对于所有输入序列,以上三个步骤肯定会执行。此外,trim_galore还支持一些其他过滤措施,以满足个性化需求。
Hardtrim5参数用于从序列的3’端切割底部,如下所示。
在: cctaagaacaagtacactccacatgcata之前
-hardtrim520: cctaagaacaagtacact序列可以通过hardt rim 5参数截断为固定长度。相应地,还有一个hardtrim3参数,它从序列的5’端开始切割碱基,如下所示。
在: caaatgttatttaagaaatggaaaat之前
-hard trim 3203360 tttttaaaaaaatggaaat软件安装起来也非常方便。首先,需要确保安装了cutadapt和fastqc,并且可执行文件位于PAH环境变量定义的路径中。然后下载trim_galore的源代码包并解压。代码如下
wget https://github.com/FelixKrueger/TrimGalore/archive/0 . 5 . 0 . tar . gz
Tarxzvf0.5.0.tar.gz在软件的安装目录中有一个名为trim_galore的可执行文件。
对于单端测序数据,基本用法如下
trim _ galore-quality 20-aagatcggaggc-length 20-out _ dirinput . FQ对于双端时序数据,基本用法如下
Trim_galore-payed-quality 20-aagatcggagc-a2 agatcggagagc-length 20-out _ dirr1.fq.gzr2.fq.gz,看完上面的内容,你知道怎么用trim _ galore过滤NGS数据的质量吗?如果您想学习更多技能或了解更多相关内容,请关注行业资讯频道,感谢您的阅读!
内容来源网络,如有侵权,联系删除,本文地址:https://www.230890.com/zhan/80650.html
